mRNAの塩基配列はリボソームによってアミノ酸配列へと翻訳され、タンパク質が生合成される。

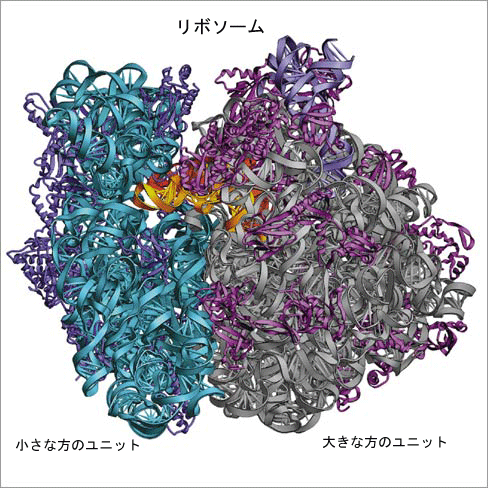
リボソームはRNA(rRNA)と70近いタンパク質で構成されている。 原核生物のリボソームは沈降係数70Sで、50Sと30Sの二つのサブユニットから成る。 50Sサブユニットは5S、23Sの二つのrRNAと30以上のタンパク質からなる。 30Sサブユニットは16SのrRNAと20以上のタンパク質からなる。
真核生物のリボソームは沈降係数80Sで、60Sと40Sの二つのサブユニットから成る。 60Sサブユニットは28S、5.8S、5SのrRNAと40以上のタンパク質からなる。 40Sサブユニットは18SのrRNAと30以上のタンパク質からなる。
リボソームの小サブユニットがまずmRNAに結合し、大サブユニットが次いで結合して翻訳が始まる。
タンパク質をもととなるアミノ酸はtRNA(transfer RNA)に結合した形(アミノアシル化tRNA)で供給される。 tRNAのアンチコドンとmRNAのコドンが相補的に結合することでmRNAの塩基配列がアミノ酸配列へと翻訳される。

リボソームが翻訳を開始する位置をどう決めているのかはまだ正確には分かっていない。 原核生物ではシャイン・ダルガノ配列(SD配列)が開始コドンの上流に共通に見られており、これと相補的な領域が16SrRNAに存在する。 原核生物の場合、SD配列を見つけてその近傍にある開始コドン(ATG)からがOpen Reading Frame (ORF)として見つけることができる。 原核生物の場合、一つのmRNAには複数のORFが存在することが多い。これをポリシストロン性(polycistronic)と呼ぶ。
一方真核生物の場合、開始コドンの上流に定まった配列が存在していないので、mRNA中のいずれのATGが開始コドンとして認識されてるのかはまだわかっていない。 真核生物のmRNAの5'末端は7メチルグアノシン(7mG)で修飾されており、これを認識してリボソームが結合すると考えられている。 5'末端から順に塩基配列を走査して最初に見つかったATGから翻訳が始まると考えられている。

mRNA中のORFは3塩基が一つのアミノ酸と対応する。 3塩基の組み合わせは64通りあり、そのうち3種類(TGA、TAA、TAG)は終止コドンを構成する(翻訳が止まる)。 残り61で20種のアミノ酸を指定している。 コドンとアミノ酸の対応はほぼすべての生物で保存されているため、生物の単一起源説を支持するものとなっている。
|
|
|
|
||||||||||||||||
|
|
|
|
||||||||||||||||
|
|
|
|
||||||||||||||||
|
|
|
|
塩基配列が翻訳されてどのようなアミノ酸配列が生じるかは三塩基ずつコドン表に従ってアミノ酸配列に置換してみればよい。 試しに「ATGGGTCGATTAGAA」という塩基配列をアミノ酸配列に置換してみる。
問題: 1文字ずらしてTGGから翻訳するとどのようなアミノ酸配列になるか検討せよ。 同様にGGGから翻訳するとどのようになるか調べよ。
長い塩基配列の場合、手動で変換するのは難しい。 そこでコンピュータプログラムを使ってみる。
遺伝子の構造(塩基配列)を決定した後、それがコードするタンパク質の構造を推測する。 しかし、塩基配列のどこから翻訳が始まっているのかはわからない。 そこで、全ての読みわく(フレーム)を調べ最も長いORFを見つける必要がある。
今回はNCBIが提供するORF finderを使用してORFを調べる。


次の塩基配列のORFを見つけよ。ORFの方向、長さ、フレームについて検討せよ。