ゲノム編集した植物のシークエンスの解析
提供: 鈴木研Wiki
(版間での差分)
(→[4] 変異型アリルの同定(ゲノム編集した植物のシークエンス解析)) |
(→[4] 変異型アリルの同定(ゲノム編集した植物のシークエンス解析)) |
||
| 46行: | 46行: | ||
==[4] 変異型アリルの同定(ゲノム編集した植物のシークエンス解析)== | ==[4] 変異型アリルの同定(ゲノム編集した植物のシークエンス解析)== | ||
'''(1) キャピラリーシークエンスを行う''' | '''(1) キャピラリーシークエンスを行う''' | ||
| − | * [3]で用いたPCR産物(制限酵素処理"前"のもの)を用いて、シークエンス解析を行う | + | :* [3]で用いたPCR産物(制限酵素処理"前"のもの)を用いて、シークエンス解析を行う |
| − | * キャピラリーシークエンスの方法は、「[[BigDye Terminaterを使ったシークエンス]]」を参照 | + | :* キャピラリーシークエンスの方法は、「[[BigDye Terminaterを使ったシークエンス]]」を参照 |
| − | ::* シークエンス反応時に使うプライマーは、[3]のPCRで使用した両側のプライマー | + | :::* シークエンス反応時に使うプライマーは、[3]のPCRで使用した両側のプライマー |
| − | ::::=PCR産物1個につき、シークエンスは2サンプル、となる (両側のプライマー=センス、アンチセンスの2種類で読むため | + | :::::=PCR産物1個につき、シークエンスは2サンプル、となる (両側のプライマー=センス、アンチセンスの2種類で読むため |
| − | ::* 1反応 (計10ul) あたりに、PCR産物を "1ul" 使用する | + | :::* 1反応 (計10ul) あたりに、PCR産物を "1ul" 使用する |
| − | :::(プライマーダイマーがある場合は、精製・定量したうえで、プロトコール通りの量を使用 (精製方法:[[NucleoSpin Gel and PCR Clean-upを使ったDNA精製]]) | + | ::::(プライマーダイマーがある場合は、精製・定量したうえで、プロトコール通りの量を使用 (精製方法:[[NucleoSpin Gel and PCR Clean-upを使ったDNA精製]]) |
'''(2) 解析に使うデータ(ファイル)を準備する''' | '''(2) 解析に使うデータ(ファイル)を準備する''' | ||
| − | * キャピラリーシークエンサーで読んだ波形ファイル(拡張子.ab1) ※上記(1)で得られたデータ | + | :* キャピラリーシークエンサーで読んだ波形ファイル(拡張子.ab1) ※上記(1)で得られたデータ |
| − | * ゲノム編集した「遺伝子のもとの配列」の情報(sgRNA配列の位置がわかるもの) | + | :* ゲノム編集した「遺伝子のもとの配列」の情報(sgRNA配列の位置がわかるもの) |
| − | + | ||
'''(3) シークエンスデータを解読して配列を決定し、アリルを同定する''' | '''(3) シークエンスデータを解読して配列を決定し、アリルを同定する''' | ||
| − | * シークエンスデータの解読方法については、「[[]]」を参照 | + | :* シークエンスデータの解読方法については、「[[]]」を参照 |
* | * | ||
* | * | ||
[[ゲノム編集した植物のシークエンスの解析:シーケンスアセンブリソフトウェア「ATGC」を使う場合]] | [[ゲノム編集した植物のシークエンスの解析:シーケンスアセンブリソフトウェア「ATGC」を使う場合]] | ||
2021年11月10日 (水) 13:53時点における版
(注意:編集中!) CRISPR-Cas9でゲノム編集した植物のシークエンスを解析し、変異を同定する方法についての解説。
目次 |
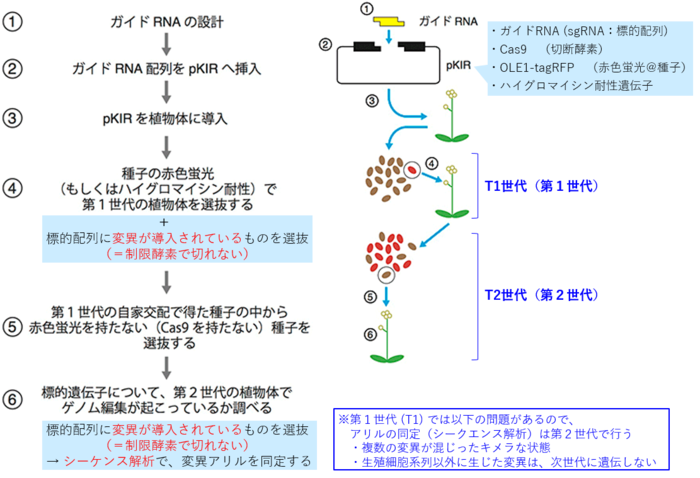
[1](概要)ゲノム編集で変異体を作成する手順:pKIベクターを用いた場合
- 以下の説明は、pKIベクターの解説サイトの図に一部加筆したもの https://www.jst.go.jp/pr/announce/20161117-2/index.html
- (※ ガイドRNAの設計方法については、別記参照)
アグロバクテリウムの感染後、シークエンス解析に至るまでの流れ (概要説明) 1) 形質転換されたT1植物を選抜し、生育する - pKIベクターを用いた場合、T1種子の中から「OLE-tagRFPが光る」ものを播いて育てる (=T-DNAが組み込まれている)
2) 目的の遺伝子がゲノム編集されている個体を選抜・生育し、T2種子をとる
- genotyping:PCR+制限酵素処理で、変異遺伝子を持つ個体を選抜 (制限酵素処理で、切れる:野生型(WT型)、切れない:変異型)
3) T2世代で、Cas9を持たない植物を選抜する - pKIベクターを用いた場合、T2種子の中から「OLE-tagRFPが光らない」ものを播いて育てる (=T-DNA、Cas9を持たない)
4) T2世代で、目的の遺伝子がゲノム編集されている個体を選抜する - 2) と同様のgenotypingを実施
5) キャピラリーシークエンサーでの解析 - 4) のgenotypingの時のPCR産物(制限酵素処理する前のもの)を、キャピラリーシークエンサーで解析する
- PCRに用いた両端のプライマー各々で解析したファイルを作成。このファイルを用いて以降の解析を行う
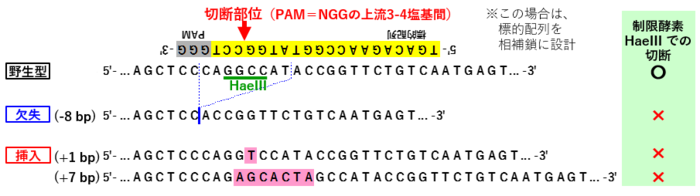
[2] ゲノム編集によってできる変異型のアリルについて
- CRISPR-Cas9によるゲノム編集では、PAM配列(NGG)の3塩基手前が切断される
- その切断個所を修復する際に、「挿入・欠失 (indel)」が生じて、フレームシフト等の変異型アリルができる
[3] 変異型アリルの検出(遺伝子型の同定:genotyping)
- ゲノム変種による変異の有無を検出できるように、制限酵素サイトの近傍にsgRNAを設計する (変異が生じると制限酵素サイトが無くなる)
- sgRNAの配列と検出に用いた制限酵素の例、については上記[2]の図を参照
- この例では、383bpのPCR産物をHaeIII処理すると、以下の長さの断片になる
- ・(野生型)制限酵素(HaeIII)で切れる -> 272bp, 111bp の2本の断片
- ・(変異型)制限酵素(HaeIII)で切れない -> 約383bp の断片
- 遺伝子型の同定を行う際の「DNA抽出、PCR、制限酵素処理」の手順については、「シロイヌナズナの遺伝子型の判定」を参照
[4] 変異型アリルの同定(ゲノム編集した植物のシークエンス解析)
(1) キャピラリーシークエンスを行う
- [3]で用いたPCR産物(制限酵素処理"前"のもの)を用いて、シークエンス解析を行う
- キャピラリーシークエンスの方法は、「BigDye Terminaterを使ったシークエンス」を参照
- シークエンス反応時に使うプライマーは、[3]のPCRで使用した両側のプライマー
- =PCR産物1個につき、シークエンスは2サンプル、となる (両側のプライマー=センス、アンチセンスの2種類で読むため
- 1反応 (計10ul) あたりに、PCR産物を "1ul" 使用する
- (プライマーダイマーがある場合は、精製・定量したうえで、プロトコール通りの量を使用 (精製方法:NucleoSpin Gel and PCR Clean-upを使ったDNA精製)
(2) 解析に使うデータ(ファイル)を準備する
- キャピラリーシークエンサーで読んだ波形ファイル(拡張子.ab1) ※上記(1)で得られたデータ
- ゲノム編集した「遺伝子のもとの配列」の情報(sgRNA配列の位置がわかるもの)
(3) シークエンスデータを解読して配列を決定し、アリルを同定する
- シークエンスデータの解読方法については、「[[]]」を参照