シロイヌナズナの遺伝子型の判定
提供: 鈴木研Wiki
2022年9月7日 (水) 17:14時点における157.110.120.101 (トーク)による版
試薬
- 0.4M NaOH
- 0.1M Tris-HCl pH 6.8
DNA抽出 マルチビーズショッカー
- 葉を一枚切り取り、サンプル破砕用チューブに入れる
- ビーズを一粒入れる
- 40 µlの0.4M NaOHを入れる
- 2000rpmで5秒オン、2秒オフのサイクルを2回行う
- 遠心(フラッシュ)
- 200 µlの0.1M Tris-HClを加える
- よく混合し、遠心(フラッシュ)
- 上清を鋳型として用いる
PCR
反応液の組成
| 1サンプル | |
| ExTaq | 0.1 µl |
| 10×ExTaq Buffer | 2 µl |
| 2.5mM dNTP | 1.6 µl |
| プライマー | 終濃度0.2 µM |
| プライマー | 終濃度0.2 µM |
| DNA | 1 µl |
| 滅菌水 | up to 20 µl |
| 合計 | 20 µl |
反応
- 95℃ 3分
- [95℃ 45秒、(アニーリング)55℃ 45秒、(伸長反応)72℃ 2分] のサイクルを35回
- 72℃ 5分 (→ 4℃ ∞)
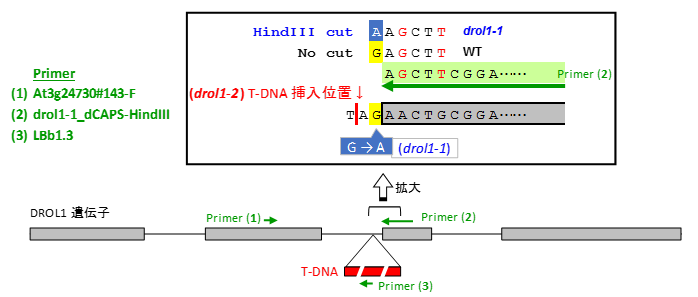
<drol1変異体の判別方法>
| 判定する 遺伝子型 |
プライマーの組み合せ | アニーリング温度 | 伸長時間 | PCR後の バンドの長さ |
備考 | |
|---|---|---|---|---|---|---|
| [A] | drol1-1 | ・At3g24730#143-F ・drol1-1_HindIII |
55℃ | 1 min でOK | (185 bp) | このPCR産物をさらに制限酵素 Hind III で切って判別 ※方法は下記参照 HindIII処理後、(WT)185 bp (drol1-1)152 + 33 bp |
| [B] | drol1-2 | ・At3g24730#143-F ・drol1-1_HindIII ・LBb1.3 |
55℃ | 2 min | 「WT」 185 bp 「T-DNA = drol1-2」 ≒355 bp |
プライマー3種類を混ぜて、「WT」「T-DNA = drol1-2」の判別を1回のPCRで判定 ※各々の増幅プライマーの組み合せ ・「WT」:At3g24730#143-F, drol1-1_HindIII ・「T-DNA = drol1-2」:At3g24730#143-F, LBb1.3 |
| [C] | drol1-1, drol1-2 ※同時に判別する場合 |
・At3g24730#143-F ・drol1-1_HindIII ・LBb1.3 |
55℃ | 2 min | 「WT」「drol1-1」 185 bp 「T-DNA = drol1-2」 ≒355 bp |
上記の[A][B]を組み合わせた検出方法 PCR後に制限酵素 Hind III で切って判別 HindIII処理後、(WT)185 bp (drol1-1)152 + 33 bp (drol1-2)≒355 bp |
| [D] | drol1-2 ※old ver. 上記推奨 |
「WT」 ・At3g24730#143-F ・anti6_At3724730 「T-DNA = drol1-2」 ・At3g24730#143-F ・LBb1.3 |
55℃ | 2 min | 「WT」 371 bp 「T-DNA = drol1-2」 ≒355 bp |
各個体について、「WT」「T-DNA = drol1-2」の2種類のPCRを行った結果を合わせて判断。 このプライマーでは、「WT」「T-DNA = drol1-2」のバンドの長さの差が小さいため、上記[B]のようなプライマー3種類を混ぜたPCRでは判別できない |
<drol1-1変異体の判別(Hind III 処理)>
| 1サンプル | |
| 滅菌水 | 5.7 µl |
| 10xM Buffer | 1 µl |
| Hind III | 0.3 µl |
| PCR産物(上記参照) | 3 µl |
| 合計 | 10 µl |
MultiNAで泳動し、バンドの長さを判別
※Hind III処理後、(WT)185 bp (drol1-1)152 + 33 bp